非結核性抗酸菌症に対する迅速・正確な病原体同定手法を開発(中村研がEmerging Microbes & Infections誌に発表)
大阪大学 微生物病研究所 松本悠希特任研究員(常勤)と中村昇太特任准教授(常勤)らの研究グループは、琉球大学大学院医学研究科の金城武士助教らと共同で、少量のデータで非結核性抗酸菌(NTM; Non-Tuberculous Mycobacteria)の菌種を迅速・正確に見分ける(同定する)手法を開発しました。
NTMとはマイコバクテリウム属細菌(※1)のうち結核菌群やらい菌を除いた種の総称であり、人の肺や皮膚にNTM症(※1)を引き起こす病原体として知られています。菌種・亜種ごとに治療方針が異なるため、適切な治療を行うためには亜種レベルでの正確な同定が必要となりますが、多種多様なNTMの亜種を正確に同定できる方法はこれまで存在していませんでした。
今回、中村特任准教授らの研究グループは、大規模なゲノムシーケンス(図1)によってNTMのゲノムデータベースの拡張、さらにMLST (Multi-Locus Sequence Typing) (※2)と呼ばれる、ゲノム配列を比較して同定を行う手法とあわせて用いることで、正確(図2)かつ迅速(図3)にNTMを同定する手法を開発しました。今回の研究成果により、10分程度のシーケンスで得られるごく少量のデータから、175種にも及ぶNTMの正確な同定が可能となりました。また、NTM症患者から分離されたNTMについて、従来の手法では同定できなかった16株を含む29株すべてを亜種レベルまで同定することに成功しました。
本研究成果により、NTMが正確に同定されることで、NTM症患者は病原体に応じた適切な治療を受けられるようになることが期待されるとともに、同定にかかる時間が劇的に短縮される可能性があります。これにより、NTM症の早期発見による予防や新たな治療方法の確立へ貢献することが期待されます。

図1)NTM同定のワークフロー。MinIONシーケンサー(図中央)を用いてゲノム配列を解読し、同定を行う。
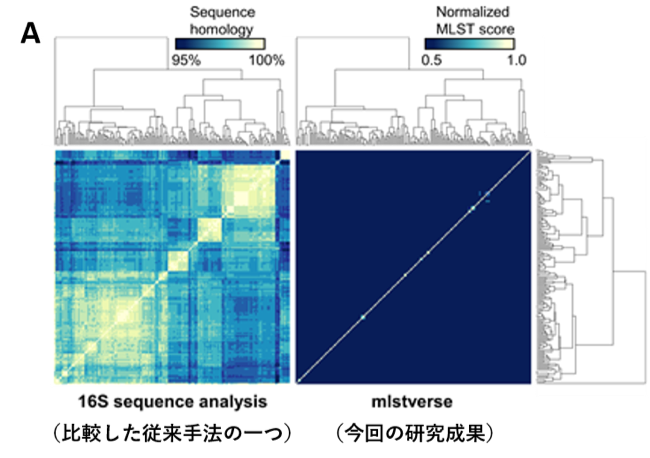
図2A)従来手法(左)と今回開発した同定法(右)の比較。白い点が相同性の高いスコア、つまりターゲットとなる菌種である可能性が高いことを表す。従来法では複数の種に対して高いスコアを示したのに対し、今回開発した手法ではターゲットとなる種に対してのみ高いスコアを示した。
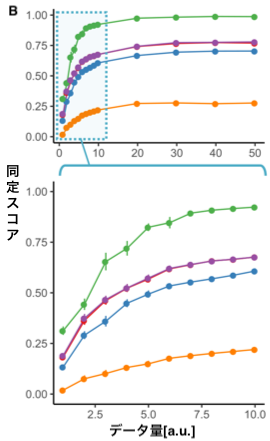
図2B)データ量と同定スコア。今回開発した手法ではX軸の値が低い位置でも同定スコアがプラトーに達しており、極めて少ないデータ量で菌種を同定できていることがわかる。
(図2ABは論文中より抜粋)
本研究成果は、2019年7月9日(火)に英国科学誌「Emerging Microbes & Infections」(オンライン)に掲載されました。
タイトル:“Comprehensive subspecies identification of 175 nontuberculous mycobacteria species based on 7547 genomic profiles”
著者名:Yuki Matsumoto, Takeshi Kinjo, Daisuke Motooka, Daijiro Nabeya, Nicolas Jung, Kohei Uechi, Toshihiro Horii, Tetsuya Iida, Jiro Fujita, Shota Nakamura
※1 NTM(非結核性抗酸菌)
マイコバクテリウム属細菌はグラム陽性細菌に分類される真正細菌の一属で、結核菌など約200種が登録されています。このマイコバクテリウム属の細菌は抗酸菌と総称され、そのうち結核菌群および、らい菌を除いた細菌を非結核性抗酸菌(NTM)といいます。これらにより引き起こされる感染症はNTM症と呼ばれ、免疫不全患者だけでなく健常者へも感染し、感染後は自覚的な症状がほとんど無いまま長い時間をかけて病状が進行します。発症後は咳・痰・血痰・発熱・食欲不振・体重減少・全身倦怠感などが見られ、抗生物質も効きづらいため、長期の適切な薬剤治療が必要となる厄介な病気です。
※2 MLST(Multi-Locus Sequence Typing)
データベースに登録された配列と同定したい検体の配列を照合することによりマッチする菌種を探す手法はsequence typingと呼ばれ、それを複数遺伝子に拡張したものが MLST と呼ばれます。菌種固有の遺伝子のDNA配列をデータベースに登録しておくことで極めて高い精度で同定が行え、データベースの規模が大きいほど検出能力が上昇します。今回、本研究においてはリボソーム分子の構成に関わる遺伝子やNTMの抗生物質耐性に関わると考えられている184遺伝子を選んだ上で、公共データベースおよび我々が新規に解読した計175種のNTMのゲノム情報を用いることで、マイコバクテリウム同定のためのデータベースを完全に新規に作成しました。








