バイオインフォマティクスセンター ゲノム情報解析分野/Standley研究室
ゲノム情報解析分野では、バイオインフォマティクスによるアプローチにより、TCR/BCRレパトアや、タンパク質-核酸相互作用、タンパク質または核酸配列の多重配列アラインメントなど、生物学的実験が困難な研究テーマを対象に、大型計算機を駆使した研究を展開しています。
多重配列アラインメント
タンパク質や核酸の比較解析において、多重配列アラインメントは重要なステップです。私たちの開発しているMAFFTプログラムは、多重配列アラインメントプログラムとして最も広く使われているものの一つです(文献1)。2002年に最初のバージョンを公表して以来、正確さと計算速度の向上のため、また、色々なタイプの新しい問題に対応するために、継続的に改良を行ってきました。例えば、ncRNAやタンパク質の立体構造を考慮したアラインメント、系統樹推定のための相互作用的な配列選択などに対応してきました(文献2)。後者は本研究室において構造モデル解析の中心的な役割を担っています。
B細胞およびT細胞受容体の特異性とレパトアの解析
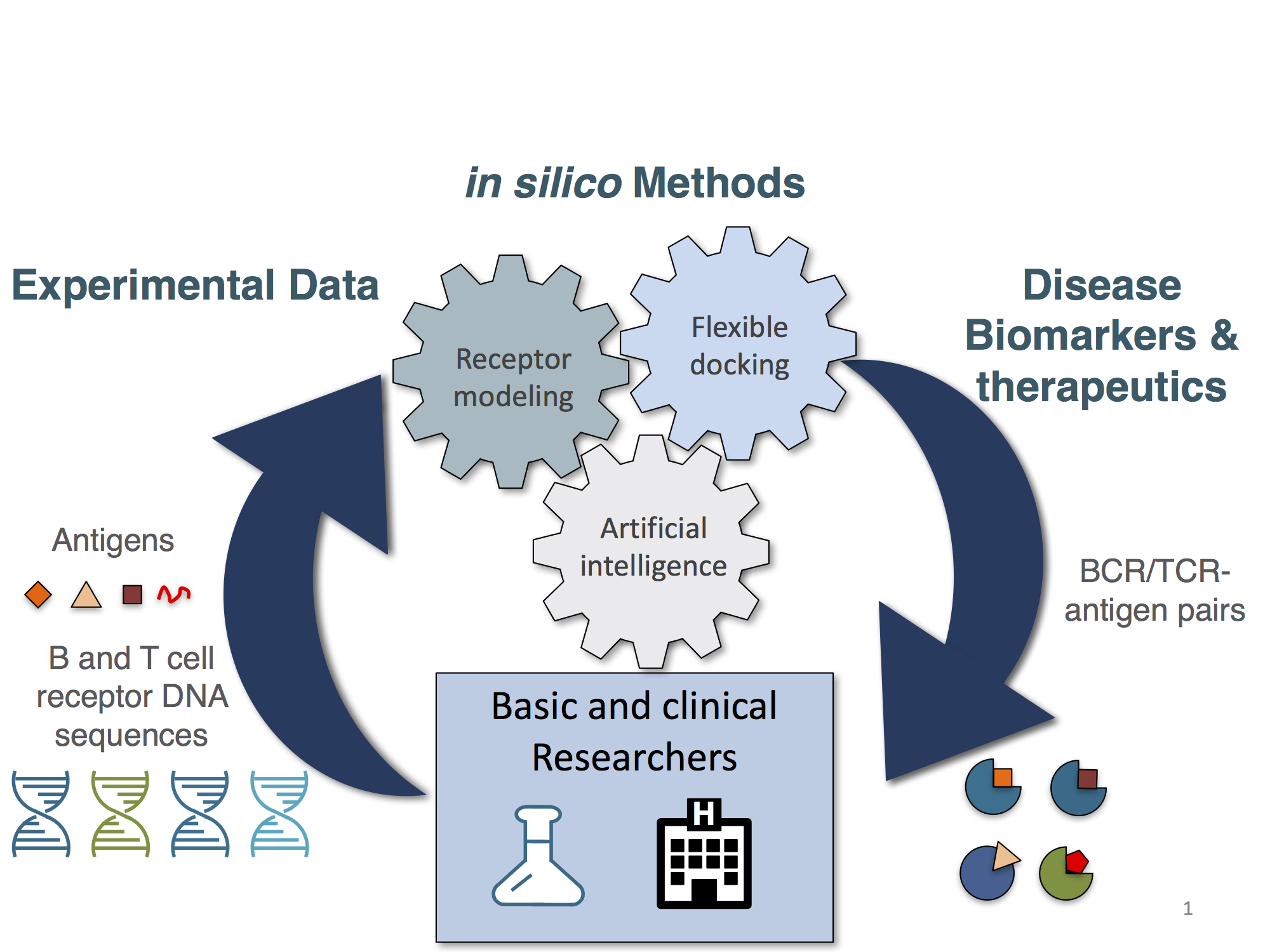
配列からのBおよびT細胞受容体抗原特異性の予測は現在重要かつ未解決の問題です。 私たちの研究室は、BおよびT細胞の受容体配列決定、構造モデリングおよびAIの組み合わせを利用してこの課題に取り組んでいます。 私達は、ハイスループットかつ正確にBCRおよびTCRの 3D構造モデルを生成するためのツールを開発しました(文献3)。 さらにこの技術を拡張して、そのような構造モデルを、その抗原およびエピトープ特異性に従ってクラスタリングできるようにしました(文献4)。 我々はまた配列情報からTCR-エピトープ-MHC構造モデルを構築するためのツールを開発するとともに(文献5)、構造情報を利用する新しいBCRエピトープ予測法の開発に取り組んでいます。 この研究の当面の目標は、特定の疾患に関連する抗原およびエピトープを、これらを認識するB、T細胞受容体と共に同定することです。
BおよびT細胞受容体の配列決定
正常な免疫システムはBおよびT細胞の広大なレパトアを持つことで、広範囲の分子を認識することができます。BおよびT細胞受容体は、異なるmRNA転写物によってコードされている2つの極めて可変性の高いポリペプチド鎖の組み合わせによって生成されます。各ポリペプチドの高い可変性と2つの異なる分子の組み合わせにより、各個人の受容体のレパトアは一般的に固有のものとなります。いくつもの免疫学的および分子的手法を用いて免疫レパトアは研究されてきましたが、これらは分解能が低いものでした。それにもかかわらず、次世代シークエンシング(NGS)の出現は、1つのサンプル中の数百万の免疫受容体配列を分析することを可能にしました(バルクシークエンシング)。これは免疫レパートリーの研究にとって大きな価値がありますが、受容体配列の組み合わせを明らかにすることはできません。この数年の間に、一細胞シークエンシングが出現し、何千もの個々のB細胞およびT細胞からの対をなすポリペプチド鎖を研究することが可能になりました。私たちは現在、健康と病気の免疫細胞レパトアを研究するために、バルクと一細胞の両方のシーケンス技術を利用しています。
タンパク質-核酸相互作用
タンパク質と核酸の相互作用は、全生命体において生物が持つ情報の流れを支配しています。免疫系でも、遺伝子の発現制御を担うDNA結合タンパク質は極めて重要な機能を果たしています。更に最近では、RNA結合タンパク質(RNA-binding proteins, RBPs)が免疫応答の強さや持続期間を決めるだけでなく、恒常性維持にも重要な役割を果たしていることが明らかになりました(文献6)。研究室では、タンパク質の核酸結合部位を予測するツールを開発し、多様なタンパク質-核酸相互作用の解析を通じてRBPによる免疫制御機構の解明を目指し研究を行っています。
メンバー
- 教授: Daron M. Standley
- 准教授: 加藤 和貴
- 准教授: Songling Li
- 特任准教授:Soyoung Park(兼)
- 特任助教: Arthur R. Millius
- 特任助教: Hedra Saputra Ismato
ホームページ
最近の代表的な論文
- (1) MAFFT multiple sequence alignment software version 7:improvements in performance and usability. Katoh, K.,et al., Mol Biol Evol (2013) 30(4) 772-80
(2) MAFFT-DASH: integrated protein sequence and structural alignment. Rozewick J., et al. Nucleic Acids Research (2019) 47(1)5-10
(3) Repertoire Builder: High-throughput structural modeling of B and T cell receptors. Schritt D., et al. Mol.Syst Des.Eng. (2019) 4,761-768
(4) Functional clustering of B cell receptors using sequence and structural features. Xu Z., et al. Mol. Syst.Des. Eng. (2019) 4, 769-778
(5) Structural Modeling of Lymphocyte Receptors and Their Antigens. Li S., et al. Methods Mol Biol. (2019) 2048:207-229
(6)Regnase-1 and Roquin Regulate a Common Element in lnflammatory mRNAs by Spatiotemporally Distinct Mechanisms.Mino, T.,et al. Cell (2015)161, 1058-1073