環境応答研究部門 システム生命医科学分野/神元研究室
我々生物の体は多様な細胞や分子で構成され、その精密な制御が複雑な生命機能を支えています。細胞や組織が形作られ、維持されるしくみを解明することは基礎生物学の根幹の問いであり、またその理解に基づく遺伝子や細胞の操作は、疾患の治療や生命工学といった応用研究にも欠かせません。
システム生命医科学分野では細胞や遺伝子が作る生物の制御回路を、膨大で複雑なデータから解き明かすことを目指しています。オミクスや1細胞計測データを統合し、コンピュータ上 (in silico)で注目する生命現象を再現するモデルを構築します。構築したモデルを通じて、複雑な生命制御のルールを解読し、さらに予測シミュレーションモデルとして活用する”in silico 生命科学研究”を行い、基礎生物学・医学・生命工学を推進することを目指します。
疾患研究の具体的な研究課題としてはがんや老化の疾患、免疫、感染症の研究課題などに取り組みます。
疾患・発生の遺伝子プログラムのin silico再構築と予測モデルとしての活用
近年、様々な実験技術・計測技術の発達により様々な生物・医学データの取得が可能になりました。一方、その複雑・膨大なデータの背後にあるメカニズムを理解し、医療に活用することは簡単ではありません。例えば、我々の体の設計図であるゲノム配列は全塩基配列が決定されましたが、データとしてその一端を計測できても、その意味を解き明かすには至っておらず、生命計測データの意味の解釈や活用にはまだ多くの壁があります。複雑・膨大な計測データの背後にある生命の文法を解読し、活用するための”翻訳機”が求められています。また、遺伝子や分子の数は膨大なので、その相互作用を含めた複雑なネットワークやシステムの理解には、計算機の助けが必要となります。
我々は計測データを繋ぎ合わせ、コンピュータ上 (in silico)で生命の振る舞いを再現するコンピュータモデルとして構築する研究を行っています。複雑な発生過程や疾患のシステムをin silicoで再現し予測する技術は、疾患を未然に予測する予測医療や、個人の違いに合わせて医療戦略を選択する個人医療の実現に欠かせない要素です。また、計算機上でのシミュレーションは現実世界の制約を超えた大スケールでのスクリーニング(探索的な研究)や、新規分子・細胞の設計にも利用できます。これら情報技術の観点から我々の研究グループは疾患研究、生命科学研究に貢献することを目指しています。
1細胞オミクス計測、AIの融合による新規研究技術開発
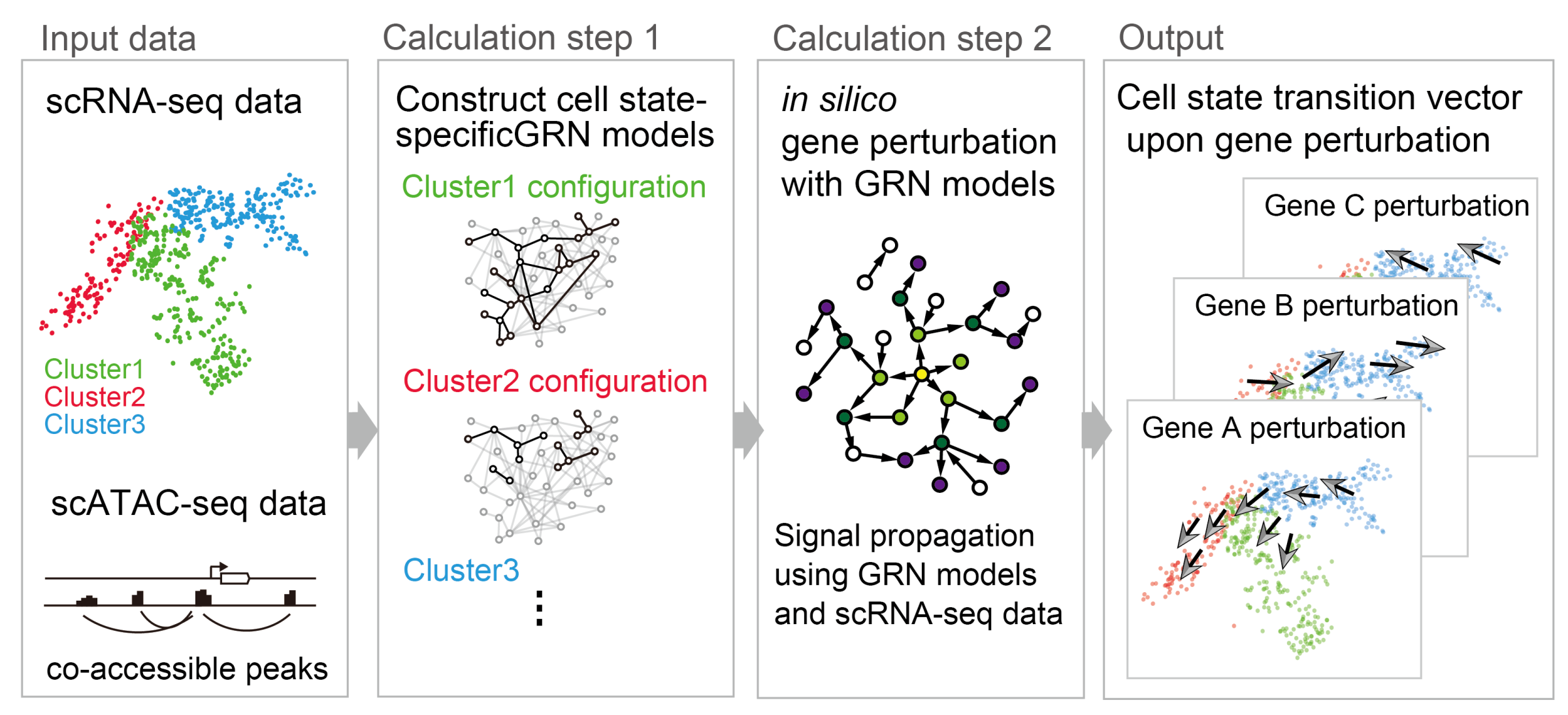
我々の体は極めて多様な細胞で構成され、発生や疾患ではその性質がダイナミックに変化していきます。我々は細胞の変化の過程を、細胞毎の不均一生や不確定性を考慮して明らかにするため、1細胞レベルでかつ網羅的な遺伝子情報を計測する1細胞オミクス解析を用いています。我々は近年、細胞の状態に特異的な遺伝子プログラムを1細胞RNA-seqデータ、1細胞ATAC-seqデータ、転写因子のDNA結合情報などを統合して遺伝子発現制御ネットワークをモデル化する技術CellOracleを開発しました(図1)。CellOracleでは作成した遺伝子制御モデルを細胞の予測シミュレーターとして利用するよう設計されており、遺伝子を変動させたときの細胞分化への影響を予測する解析を行います。我々はこの手法を用いてマウス・ゼブラフィッシュといったモデル生物での発生過程の遺伝子プログラムの研究を行ったり、ヒトでの疾患の研究に応用してきました。
オミクス計測の解析やin silico研究では、膨大なデータの中にパターンを発見し、その知見を生命の予測モデル・シミュレーションモデルへと変換するためにAI・機械学習の活用が重要になります。我々のグループでのAI研究では、単に既存のAI手法を生命データに転用するのではなく、生物の知識や仮説を積極的に取り入れたAIモデルやAI学習方法の開発を行い、生物学が組み込まれた機械学習(Biology-Informed Machine Learning)の方法論の研究に焦点を当てます。
生物情報技術を利用した生命工学・合成生物学研究
疾患の治療や、再生医療では細胞や遺伝子を操作して異常な細胞を正常な状態に戻したり、病気で失われた細胞を補うための細胞を作成する必要があります。我々はin silicoで再構築した生命現象の予測モデルを遺伝子や細胞の操作に活用します。従来のアプローチでは、リプログラミングといった細胞操作や薬剤の新規発見のためには、ランダムに効果的な条件や新規物質を試すといったランダムスクリーニングが行われます。しかし、細胞の操作や遺伝子の操作の潜在的な条件は無数に存在し、複雑な操作を行おうとするほどに、ランダムに発見できる可能性が低くなってしまいます。
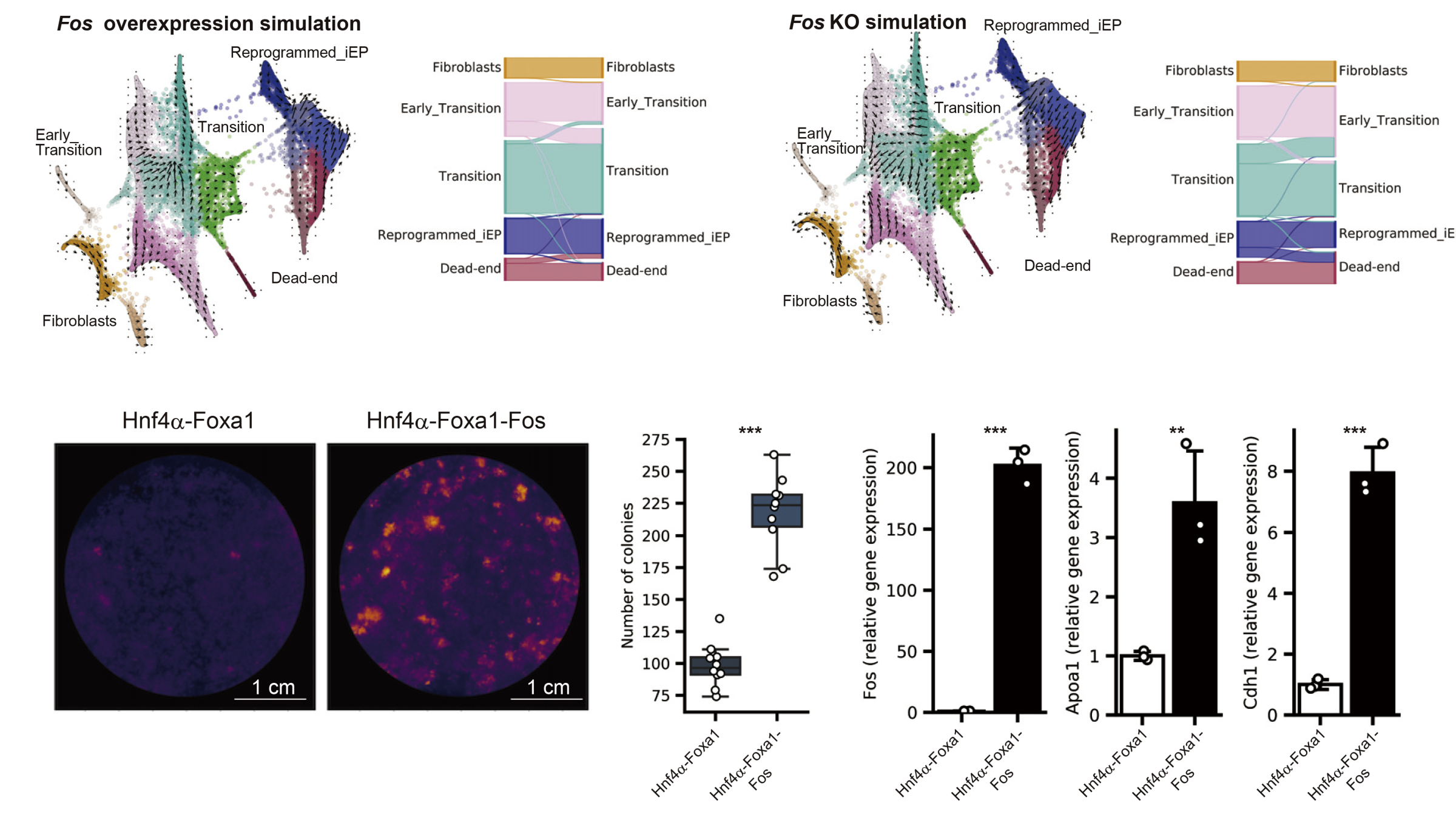
我々はランダムスクリーニングだけではなく、細胞の制御の体系的な理解と予測に基づいた意図的な細胞制御・設計法の構築が重要であると考え、研究を行っています。そのために、コンピュータ上 (in silico)で細胞や遺伝子の注目する働きを再現するAIモデルや数理モデルを構築し、その予測やシミュレーションに基づいた細胞や遺伝子の操作を行います。近年発表した論文では、肝臓や腸の元になる内胚葉前駆細胞(Endoderm Progenitor)を作成する過程にin silico解析技術を適応し、予測シミュレーションモデルを活用した手法で内胚葉前駆細胞の新たな誘導のための重要遺伝子を多数同定しました(図2)。また、情報解析技術やin silico予測技術と併用する合成生物学・DNA工学の技術開発も行っています。
メンバー
- 教授:神元 健児
- 助教:伊藤 謙治
- 特任助教:野崎 修平(兼)
- 特任助教:萩原 佑紀
ホームページ
最近の代表的な論文
1. Single-cell lineage capture across genomic modalities with CellTag-multi reveals fate-specific gene regulatory changes. Jindal, K., et al., Nature Biotechnology (2024) 42, 946–959.
2. Defining cardiac functional recovery in end-stage heart failure at single-cell resolution. Amrute, J.M. et al., Nature Cardiovascular Research. (2023) 2, 399–416.
3. Dissecting cell identity via network inference and in silico gene perturbation. Kamimoto, K.,et al., Nature (2023) 614, 742–751.
4. Gene regulatory network reconfiguration in direct lineage reprogramming. Kamimoto, K., et al., Stem Cell Reports (2023) 18, 1, 97–112
5. The coding and long noncoding single-cell atlas of the developing human fetal striatum. Bocchi, V.D., et al., Science (2021) 1979-372. 10.1126/science.abf5759,
6. Single-cell mapping of lineage and identity in direct reprogramming. Biddy, B.A., et al., Nature (2018) 564, 219–224.