バイオインフォマティクスセンター 生物情報解析分野 RNA情報学グループ/山﨑研究室
RNA情報学研究室では、特にメッセンジャーRNA(mRNA)に関する情報の解析を通じて、遺伝子発現制御の仕組みを理解し、それを応用することで社会に貢献することを目指しています。
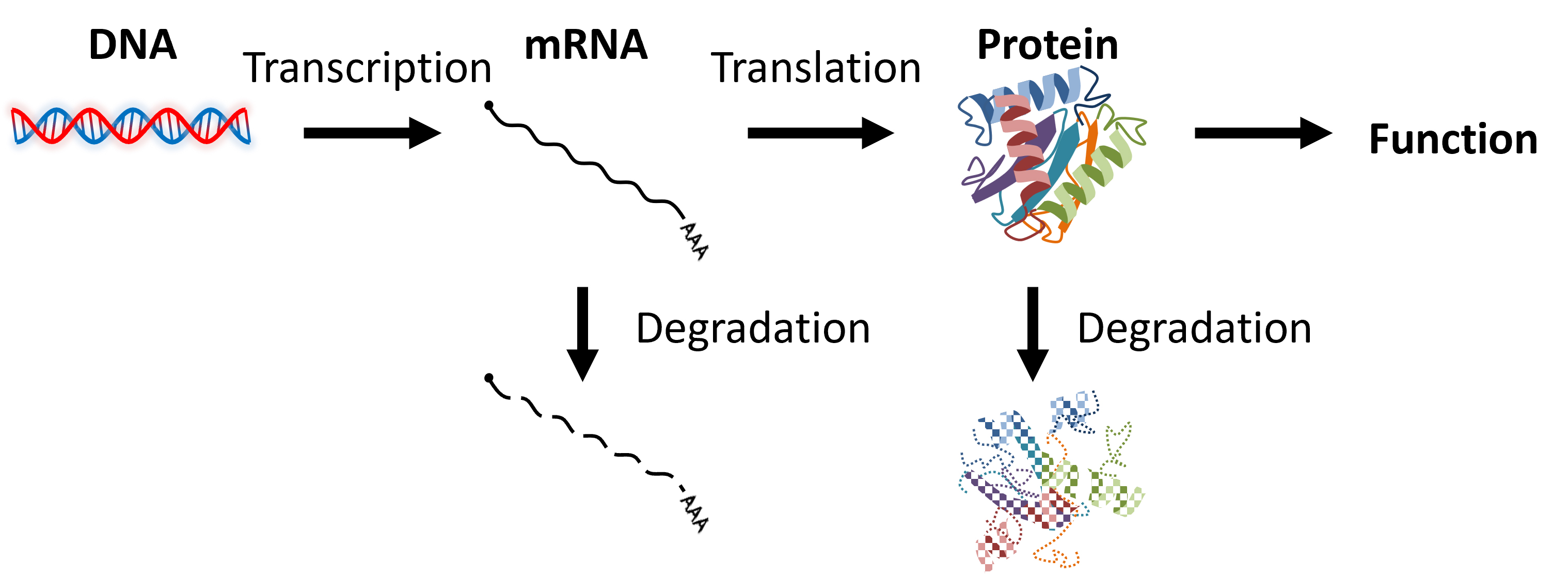
私たちの体内では、酵素や構造タンパク質をはじめとするさまざまなタンパク質が、生命活動を支える中心的な役割を担っています。その設計図はDNAに記録されていますが、DNAの情報が直接タンパク質へと変換されるわけではありません。まず、DNAの情報がmRNAに転写され、それが翻訳されることでタンパク質が合成されます(図1)。この一連の過程は「遺伝子発現」と呼ばれ、生命活動を正常に維持するためには、タンパク質が作られるタイミングや量が細胞の種類や環境条件に応じて精密に制御される必要があります。
最終的なタンパク質の発現量の制御には、mRNAの転写効率、mRNAの安定性、mRNAの翻訳効率、タンパク質の安定性が大きく関与します。先天的・後天的な要因によってこの制御が破綻すると、遺伝性疾患やがんなど、さまざまな疾患の原因となります。したがって、生命の基本的な仕組みを理解し、疾患の原因を解明するためには、遺伝子発現制御の詳細な理解が不可欠であり、その鍵を握るのがmRNAの研究です。
さらに、mRNAの研究や関連技術は、医薬品開発やバイオ産業の発展においても重要な役割を果たしています。例えば、mRNAを利用したワクチンや遺伝子治療薬は、従来の医薬品とは異なる革新的な治療手段を提供します。また、医療・産業用タンパク質の大量生産においても、mRNAは重要な設計図として機能します。mRNAの配列を最適化することは、医薬品や産業用酵素の開発・製造の効率化にもつながります。
これらの理由から、mRNAの研究は生命科学の発展のみならず、医療や産業の進歩にも不可欠な分野となっています。当研究室では、mRNAの転写、分解、翻訳に関わる複雑な制御機構を、最新のシーケンシング技術や機械学習を駆使して解明し、これらの知見を新しい医療技術や産業技術の開発に応用しています。
mRNA配列の決定機構の解明と応用
転写は遺伝子発現の最初のステップであり、その後のすべてのプロセスを決定する極めて重要な段階です。この段階では、細胞の種類や環境条件に応じて転写されるmRNAの量が精密に制御されています。しかし、それと同様に重要なのが、「どのようなmRNA配列が転写されるのか」、すなわちmRNAの質の制御です。
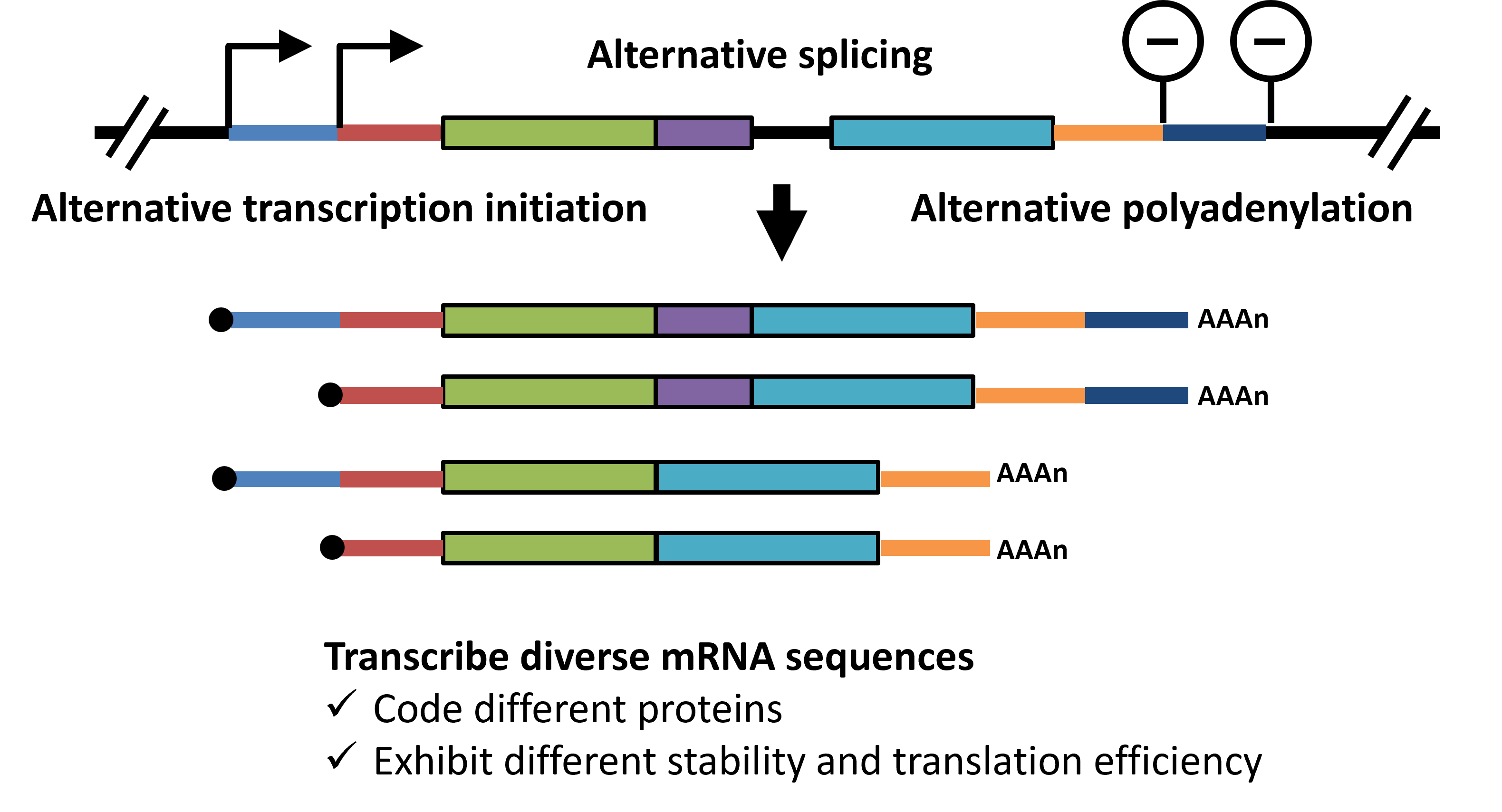
ヒトのタンパク質をコードする遺伝子の数は約2万とされ、単細胞生物であるクラミドモナス(コナミドリムシ)の約1万8千と大きな差はありません。しかし、ヒトでは選択的スプライシングをはじめとする多様な転写後調節機構が高度に発達しており、1つの遺伝子から複数種類のmRNAが転写されることで、最終的なタンパク質の種類が大幅に増加しています(図2)。さらに、選択的転写開始や選択的ポリアデニル化によって、同じタンパク質をコードしながらも、異なる安定性や翻訳効率を持つmRNAが多数転写されており、それら多様なmRNAによって非常に複雑な制御が行われています。このような選択的転写機構は、高度な遺伝子発現に必要不可欠であり、その異常が疾患の発症や進行に関与していることが明らかになってきています。
そこで、当研究室では、CAGE-seqやナノポアロングリードcDNAシーケンシングデータと機械学習を組み合わせ、mRNAの転写開始点、スプライシングパターン、ポリA付加部位の決定機構を解析し、その位置を予測するシステムの開発を進めています。これにより、選択的転写機構に影響を及ぼすDNA変異の検出や、有用タンパク質の生産における高度な遺伝子の設計を目指しています。
mRNA安定性と翻訳効率の制御機構の解明と応用
mRNAの転写効率と同様に、mRNAの安定性と翻訳効率も遺伝子発現の重要な制御段階です。例えば、転写因子は細胞内外の環境変化やシグナル伝達に迅速に応答する必要があるため、それをコードするmRNAは不安定であり、不要な条件では速やかに分解される傾向があります。一方、構造タンパク質などは、安定した高いレベルでの発現が求められるため、それをコードするmRNAは安定性が高い傾向があります。mRNAの翻訳効率は、多くの場合、タンパク質の蓄積量と相関することが知られており、最終的な遺伝子発現に与える影響は、転写段階に次いで大きいとされています。また、mRNAの翻訳効率は細胞内のエネルギー消費の調節にも関与しており、細胞の状態に応じてさまざまな生合成関連遺伝子の翻訳効率を制御しています。
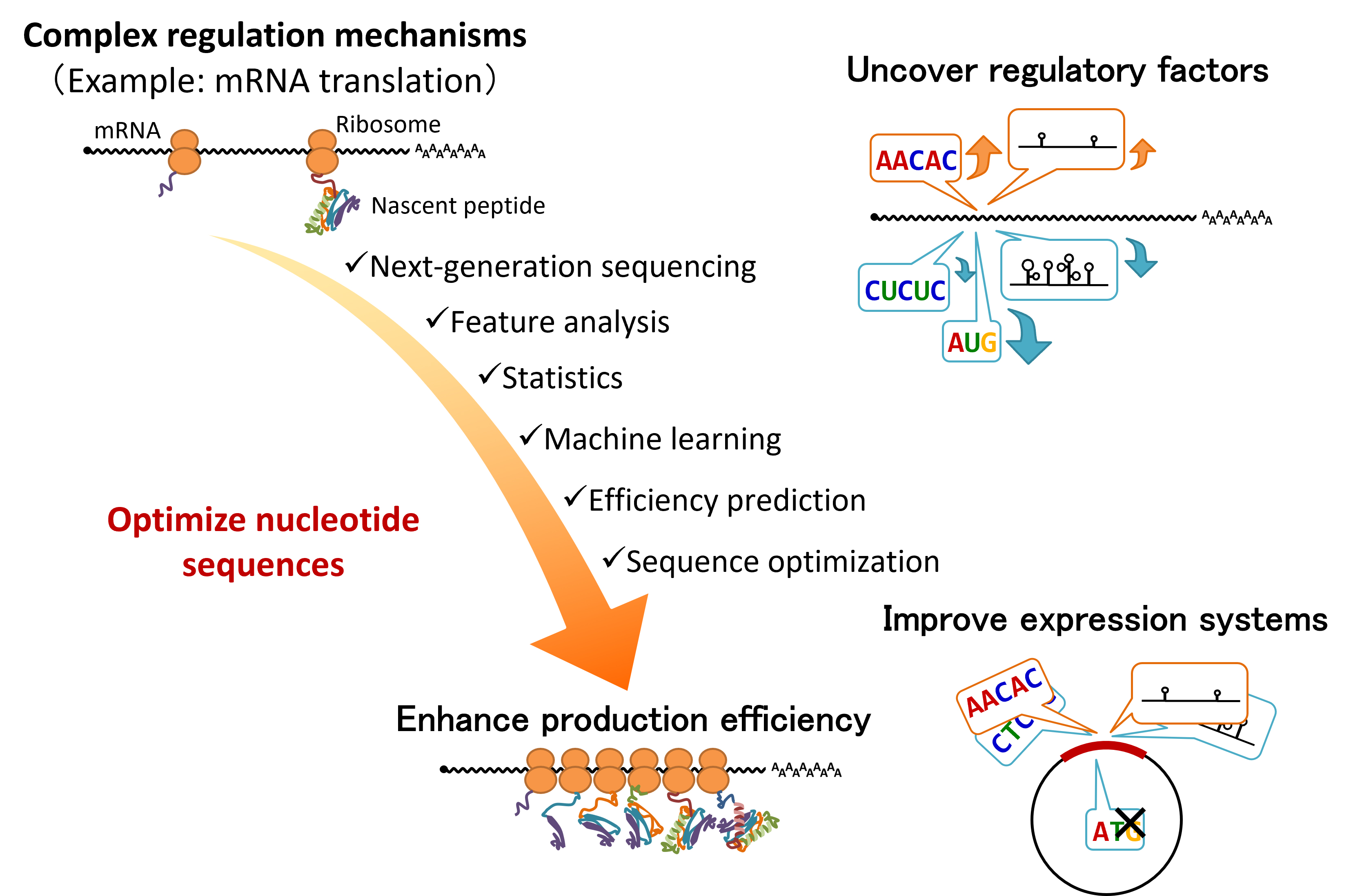
このようなmRNAの安定性と翻訳効率は、mRNAの5'非翻訳領域(5'UTR)、コーディング領域(CDS)、3'非翻訳領域(3'UTR)に存在する多様な制御配列によって決定されます。当研究室では、転写阻害剤追跡法やポリソーム分画法と、mRNAアイソフォームを詳細に評価できるCAGE-seqやナノポアロングリードcDNAシーケンシングを組み合わせることで、従来よりも高精度かつ詳細なmRNAの安定性や翻訳効率のデータを取得しています。このデータに基づき、網羅的な特徴評価、統計学的手法、機械学習法を組み合わせて、mRNAの安定性と翻訳効率の制御に関わる特徴の解明を行っています。得られたデータは選択的転写機構の情報とも統合されており、mRNAの転写、分解、翻訳の各段階をまたぐ包括的な遺伝子発現制御の研究を進めています。
また、mRNA医薬品の開発や有用タンパク質の大量生産においても、mRNAの安定性と翻訳効率の制御は極めて重要です。これらの医療やバイオ産業では、より安定し、翻訳効率の高いmRNA配列が求められています。そこで当研究室では、前述のデータや超並列レポーターアッセイ法によって取得した大規模データ、機械学習、遺伝的アルゴリズムを活用し、mRNAの配列最適化システムを開発しています(図3)。本システムは、従来よりも高品質なデータに基づく高精度な予測モデルを搭載しており、これを用いることで、持続的かつ高効率なタンパク質合成が可能となることが期待されます。
RNAの応用の促進
RNAの応用は、医療、農業、畜産業、水産業、バイオ産業などの分野で急速に拡大しています。当研究室では、RNAの安定化や翻訳効率向上を目的とした配列設計システムの開発を進めており、これらの分野における多様な応用に貢献することを目指しています。
その一例が、無細胞タンパク質合成のためのmRNA設計です。無細胞タンパク質合成とは、生きた細胞を使用せずに、抽出した細胞成分を用いてタンパク質を合成する技術です。細胞培養を必要としないため、短時間でタンパク質を生産できることが大きな利点です。また、mRNAを直接鋳型として使用できるため、遺伝子組み換え操作を行う必要がありません。当研究室では、超並列レポーターアッセイ法を用いて、無細胞タンパク質合成系におけるmRNAの安定性と翻訳効率のデータを取得し、それに基づいて最適なmRNA配列を設計するシステムの構築を進めています。この開発中のシステムにより、すでに複数のタンパク質の合成量を大幅に向上させることに成功しています。
RNAを活用した技術は、次世代の医療や産業の発展を支える重要な要素であり、私たちはその最前線で研究を進めています。
メンバー
- 准教授: 山﨑 将太朗
- 特任助教: NEVIEN KAMEL MOHAMED ABDELKHALEK
最近の代表的な論文
1.Sequence features around cleavage sites are highly conserved among different species and a critical determinant for RNA cleavage position across eukaryotes. Ueno D., et al., J. Biosci. Bioeng. (2022) 143: 450-461
2.Methods for detecting RNA degradation intermediates in plants. Ueno D., et al., Plant Sci. (2022) 318: 111241
3.Feature selection for RNA cleavage efficiency at specific sites using the LASSO regression model in Arabidopsis thaliana. Ueno D., et al., BMC Bioinformatics (2022) 22: 380
4.Identification of 5'-untranslated regions that function as effective translational enhancers in monocotyledonous plant cells using a novel method of genome-wide analysis. Yamasaki, S., et al., Plant Biotechnol. (2018) 35: 365-373
5.Arabidopsis thaliana cold-regulated 47 gene 5-untranslated region enables stable high-level expression of transgenes. Yamasaki, S., et al., J. Biosci. Bioeng. (2018) 125: 124-130
6.Changes in Polysome Association of mRNA Throughout Growth and Development in Arabidopsis thaliana. Yamasaki, S., et al., Plant Cell Physiol. (2015) 56: 2169-2180